【医学影像 AI】U-Bench: 基于 100 种变体基准测试的 U-Net 全面解析
U-Bench是首个全面评估100种U-Net变体的大规模基准测试平台,涵盖28个数据集和10种医学影像模态。该研究提出U-Score新指标,综合评估模型性能与计算效率,揭示了现有方法在统计显著性、零样本泛化能力和效率权衡方面的不足。该工作填补了U-Net变体缺乏系统性评估的空白,为临床部署和未来研究提供了重要参考。
更多内容请关注【医学影像 AI】
U-Bench: 基于 100 种变体基准测试的 U-Net 全面解析
0. 论文简介
0.1 基本信息
2025年 Tang Fenghe 等发布论文 “U-Bench: 基于 100 种变体基准测试的 U-Net 全面解析(U-Bench: A Comprehensive Understanding of U-Net through 100-Variant Benchmarking)”。
论文标题: U-Bench: A Comprehensive Understanding of U-Net through 100-Variant Benchmarking
作者: Fenghe Tang, Chengqi Dong, Wenxin Ma, Zikang Xu, Heqin Zhu, Zihang Jiang, Rongsheng Wang, Yuhao Wang, Chenxu Wu, Shaohua Kevin Zhou
论文地址: arxiv
代码仓库: github
引用格式: Tang, F., Dong, C., Ma, W., et al. (2025). U-Bench: A comprehensive understanding of U-Net through 100-variant benchmarking. arXiv preprint arXiv:2510.07041.
0.2 摘要
在过去十年中,U-Net 一直是医学图像分割领域的主流架构,催生了数千种 U 型变体。尽管应用广泛,但目前尚无全面的基准测试来系统评估这些变体的性能和实用性,这主要是由于统计验证不足,且对不同数据集间的效率和泛化能力考量有限。
为填补这一空白,我们提出 U-Bench—— 首个大规模、统计严谨的基准测试平台,可在 28 个数据集和 10 种成像模态上评估 100 种 U-Net 变体。我们的贡献主要体现在三方面:
- 全面评估:U-Bench 在三个关键维度上评估模型:统计鲁棒性、零样本泛化和计算效率。我们引入了一个新颖的指标 U-Score,它联合捕获了性能-效率的权衡,为模型进展提供了面向部署的视角。
- 系统分析和模型选择指导:我们总结了大规模评估的关键发现,并系统地分析了数据集特征和架构范式对模型性能的影响。基于这些见解,我们提出了一个模型顾问代理,以指导研究人员为特定数据集和任务选择最合适的模型。
- 公开可用性:我们提供所有代码、模型、协议和权重,使社区能够重现我们的结果并用未来的方法扩展基准。
总之,U-Bench 不仅揭示了先前评估中的不足,而且为未来十年 U-Net 类分割模型的公平、可复现且与实际相关的基准奠定了基础。
项目可通过 https://fenghetan9.github.io/ubench 访问。代码可在 https://github.com/FengheTan9/U-Bench 获取。
1. 引言
医学图像分割是一项关键且具有挑战性的任务,通过为医生提供客观、精准的感兴趣区域参考,可显著提升诊断效率(Zhou 等,2017)。过去十年间,U-Net(Ronneberger 等,2015)凭借其编码器 - 解码器结构及跳接连接,能有效融合多尺度特征,已成为医学图像分割领域的基石。基于其在多种模态下优异的分割效果,研究者们提出了大量 U 型变体以进一步提升性能,包括轻量化设计(Valanarasu 和 Patel, 2022;Tang 等,2024;Chen 等,2024a;Valanarasu 等,2021;Cao 等,2022)、注意力机制(Oktay 等,2018;Tang 等,2023)、多尺度特征融合(Zhou 等,2018;Huang 等,2020),以及近年涌现的基于 Mamba(Liu 等,2024a;Wu 等,2025b)、RWKV(Ye 等,2025;Jiang 等,2025)的架构,还有混合架构(Chen 等,2021;Tang 等,2025b;Dong 等,2025;Tang 等,2025a)。过去十年间,已有超过一万种 U-Net 变体被提出,截至 2025 年,近千项研究采用 U 型网络用于医学图像分割。
在数量庞大的 U-Net 变体中,一个核心挑战仍未解决:如何对这些变体进行公平且全面的比较?尽管已有部分基准测试和综述尝试梳理这一领域的发展(表 1),但它们大多缺乏大规模、系统性的评估。模型改进的稳健性、零样本泛化能力和计算效率等关键方面常被忽视,且未能对数据集特定特征与模型架构进行完整深入的分析。尽管近年研究报告了性能提升,但许多研究未提供统计验证(73% 的研究省略了这一步骤,图 1D),基线对比不完整,或数据集覆盖范围有限。此外,效率对于实际临床部署至关重要(Vashist, 2017;Wenderott 等,2024;Xu 等,2025),却极少被纳入考量。更严重的是,评估通常局限于域内场景(84% 的研究忽略零样本评估,图 1D),而临床实践中,不同机构和标注协议必然导致域转移(Yan 等,2019;Koch 等,2024)。这些空白使得 U-Net 变体在真实场景中的稳健性和实用性仍未得到充分验证(Niu 等,2024)。
为系统、全面地评估 U 型医学图像分割模型,我们提出 U-Bench—— 首个大规模、统计严谨且面向效率的 U-Net 及其变体基准测试平台。
U-Bench 基于三个核心维度构建:
(1)广泛的数据集与模型覆盖:我们实现了 100 种近年提出的 U-Net 变体,并在 28 个基准数据集上进行评估,涵盖 10 种不同的成像模态(超声、皮肤镜、内镜、眼底摄影、组织病理学、核成像、X 射线、磁共振成像、计算机断层扫描和光学相干断层扫描;图 1A、C)。
(2)严谨且全面的评估:所有模型均通过与基线 U-Net 的性能对比进行统计显著性计算,确保对比的稳健性与公平性(图 1E)。为体现临床实用性,我们进一步评估了跨模态的零样本泛化能力。此外,针对真实场景中边缘部署的实际需求,我们引入 U-Score 这一基于统计的大规模指标,综合考量精度、参数量、计算成本和推理速度(图 1F)。
(3)公开可获取性与可复现性:U-Bench 的模型均采用官方代码实现、预训练权重(若有)和深度监督策略(若适用)。同时,U-Bench 开放所有代码、模型和实验协议,方便科研社区复现研究结果,并将该基准测试扩展至未来的新方法。
基于这项大规模评估,我们得出了一些挑战传统认知的关键发现。IoU 等传统指标已呈现饱和迹象,区分能力有限(图 1F)。此外,报告的性能提升往往不一致或缺乏统计显著性(图 1E)。与此同时,U-Score 的上升趋势反映了研究界对存储和计算成本的关注度日益提升(图 1F)。为探究这些动态,我们对 U-Net 变体进行了系统分析,考察了数据集和架构因素在不同模态、架构类型和计算资源限制下对模型性能的影响(图 1G)。基于这些分析,我们引入了模型顾问智能体,可根据数据集和任务属性推荐合适的架构,为临床和科研从业者提供切实可行的指导。
我们的贡献可总结为:
- 我们提供了一个全面的评估基准,在 28 个数据集(涵盖 10 种模态)上对 100 种 U 型变体进行评估,涵盖统计稳健性、零样本泛化能力和计算效率三大维度。为更好地捕捉精度与效率的权衡关系,我们引入 U-Score 这一基于大规模统计分析的新型指标,实现公平、全面的评估。
- 我们总结了大规模评估的关键发现:大多数变体虽有性能提升,但很少能在域内评估中显著优于原始 U-Net;零样本性能则取得了显著且令人鼓舞的进步;U-Score 的上升趋势表明,研究重心正从单纯追求精度转向平衡精度与效率。
- 我们拆解了数据集特征和架构设计等不同因素,揭示了它们对性能和效率的影响,并进一步构建了模型推荐系统,帮助研究者在不同数据和资源条件下识别合适的架构。
- 我们开源了 U-Bench 及所有预训练权重,为医学图像分割提供了一个大规模、综合评估的基准平台,以促进社区开展公平、稳健、可泛化且高效的研究。

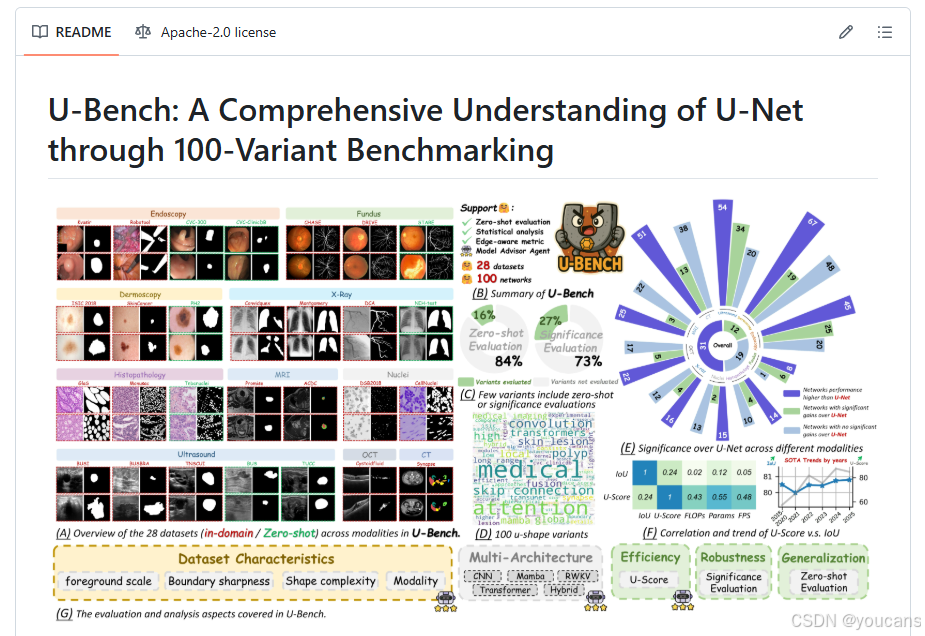
图1:U-Bench概述。
(A) U-Bench的总结,涵盖了对U形架构最全面的大规模评估。
(B) U-Bench模型库中100种已发布的U形变体的词云。
© U-Bench数据库中28个数据集的示例。红色/绿色框:用于评估的域内/零样本划分。
(D) 文献分析。在100篇近期工作中,84%的论文忽略了零样本评估,73%的论文缺乏统计显著性检验。
(E) 显著性分析。只有少数模型在U-Net上实现了统计意义上的显著提升。
(F) 新指标U-Score概述。上:IoU未考虑效率,而U-Score与分割性能和效率指标均显示出强相关性。下:尽管IoU显示出饱和趋势,U-Score突出了向更高效模型发展的年度趋势。
(G) U-Bench涵盖的评估和分析方面。
表1:U-Bench与其他医学图像分割基准的比较。详细信息可在附录B中找到。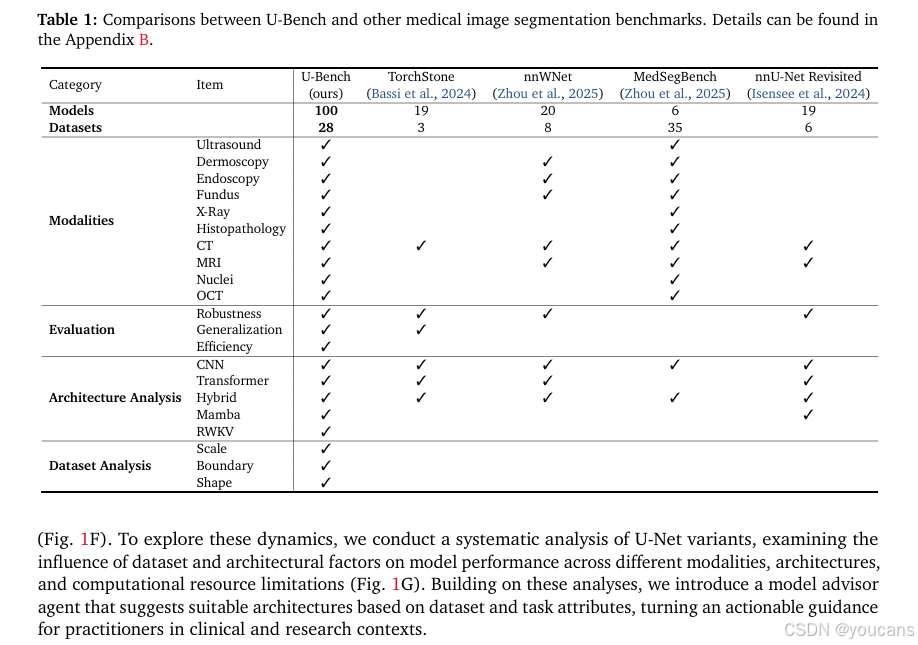
2. U-Bench 构建
为实现对 U-Net 变体的全面、公平且实用评估,我们从数据集库、模型库、评估协议与指标设计三个核心维度构建 U-Bench 基准平台。本节将详细阐述各模块的设计逻辑与实施细节。
2.1 预备知识:
U 型结构设计U 型模型通常包含三个核心组件:分层编码器、解码器、瓶颈层及跳接连接。
给定输入图像 x ∈ R 3 × H × W x \in \mathbb{R}^{3 \times H \times W} x∈R3×H×W。
- 编码器(Encoder (⋅))通过自上而下的 N 个阶段提取多尺度特征 f i f_i fi,表示为 { f i } i = 1 N \{f_i\}_{i=1}^N {fi}i=1N,其中 f i ∈ R C i × H 2 ( i − 1 ) × W 2 ( i − 1 ) f_i \in \mathbb{R}^{C_i \times \frac{H}{2^{(i-1)}} \times \frac{W}{2^{(i-1)}}} fi∈RCi×2(i−1)H×2(i−1)W。
- 瓶颈层(Bottleneck (⋅))对编码器的最后一个输出特征进行处理,
- 解码器(Decoder (⋅))由自下而上的 N − 1 N-1 N−1 个上采样阶段组成,生成解码器特征 d j d_j dj,每个阶段均包含用于特征融合的跳接连接(Skip-connection (⋅))。
- 最终预测结果 x ~ \tilde{x} x~ 由顶层解码器阶段后的分割头生成。
各类变体的差异如图 2 所示:卷积神经网络(CNN)及相关架构(注意力机制、Mamba、RWKV)构成核心构建模块,这些模块可采用纯 CNN / 注意力机制、并行或串行配置,应用于编码和解码过程。详细分类见附录 B 和附录 D。
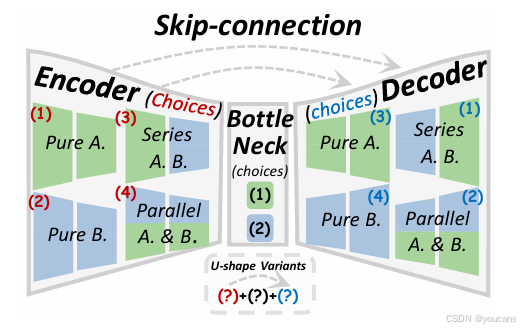
图2:U形网络的总结。
该网络包括一个编码器、一个瓶颈部分以及一个带有跳跃连接的解码器,每个部分都可以整合注意力门控和多尺度融合。
2.2 数据集库(Data Zoo)
数据集的多样性与代表性是评估模型泛化能力的关键。我们精心筛选了 28 个公开医学图像分割数据集,覆盖 10 种核心成像模态,同时兼顾域内训练与零样本泛化测试场景,确保评估的全面性与临床相关性。
- 模态覆盖:涵盖超声(US)、皮肤镜(Dermoscopy)、内镜(Endoscopy)、眼底摄影(Fundus)、组织病理学(Histopathology)、核成像(Nuclear Imaging)、X 射线(X-ray)、磁共振成像(MRI)、计算机断层扫描(CT)、光学相干断层扫描(OCT),覆盖临床常见诊断场景。
- 数据划分:20 个数据集用于域内训练与验证,8 个数据集作为零样本测试集(同模态但不同采集域 / 机构),模拟临床中设备、标注协议差异导致的域转移问题。
- 数据特征标注:对每个数据集的核心属性进行量化标注,包括前景目标尺度(小 / 中 / 大)、边界清晰度(模糊 / 中等 / 清晰)、目标形状复杂度(简单 / 中等 / 复杂),为后续架构影响分析提供基础。
2.3 模型库(Model Zoo)
我们系统收集并复现了 100 种近年主流 U-Net 变体,涵盖 5 类核心架构范式,确保模型选择的代表性与前沿性,同时严格统一实现标准以保障对比公平性。
- 架构分类:
- CNN 类:基于传统卷积的轻量化、多尺度优化变体(如 U-Net++、ResU-Net、MobileU-Net 等);
- Transformer 类:融合自注意力机制的变体(如 TransU-Net、SwinU-Net、Vision Transformer 驱动的 U 型架构等);
- Mamba 类:采用结构化状态空间模型的新兴变体(如 MambaU-Net、MedMamba 等);
- RWKV 类:基于循环神经网络与注意力融合的变体(如 RWKV-U-Net、MedRWKV-Seg 等);
- 混合类(Hybrid):结合 CNN 局部特征提取与 Transformer/Mamba/RWKV 长程依赖建模的架构(如 CNN-Transformer Hybrid、Mamba-CNN Fusion 等)。
- 统一实现标准:
所有模型采用 PyTorch 框架复现,遵循官方论文描述的网络结构、激活函数与优化策略;若无官方预训练权重,则使用统一初始化策略从头训练;一致采用交叉熵损失函数(部分数据集结合 Dice 损失),训练轮次、学习率调度策略统一优化,确保仅架构差异影响评估结果。
2.4 评估指标
参考现有研究(Luo 等,2025; Jiang 等,2025; Tang 等,2025b; Valanarasu 和 Patel, 2022; Tang 等,2024),我们采用交并比(IoU)评估分割性能。为验证模型间性能差异的统计显著性,我们通过配对样本 t 检验,将每个变体与基线 U-Net 进行对比。U-Bench 同时纳入计算效率指标,包括参数量(单位:M)、计算量(单位:G)和推理速度(FPS)。所有结果细节见附录 D。
-
零样本评估
为评估模型在训练分布之外的泛化能力,我们在相同模态和任务的未见过数据集上进行零样本推理。具体而言,模型在源数据集上完成训练后,直接在相同模态但采集域不同的未见过数据集上进行评估。详细的数据集划分见附录图 1 ©。该评估方式与临床需求一致 —— 在实际应用中,设备、机构和患者群体的差异常导致域转移现象。 -
U-Score 指标
为评估模型的实际部署价值,我们提出 U-Score 这一统一指标,综合考量分割精度与计算效率。对于每个模型 i,先计算其 IoU 分割精度 Ai、参数量 Pi、计算量 Gi 和推理速度 Si;再基于模型库中所有模型的 10 分位 - 90 分位值,将这些指标归一化至 [0,1] 区间,得到 ai、pi、gi、si。采用等权重调和平均函数ℋ(⋅) 计算效率子分数 Effi = ℋ(pi, gi, si),确保无单一因素主导评估结果。最终 U-Score 定义为 U-Scorei = ℋ(ai, Effi),平等融合分割精度与计算效率,既奖励在精度 - 效率权衡中表现优异的模型,也为 IoU 提供了面向部署的替代评估方案。更多细节见附录 E。
计算逻辑如下:
- 指标归一化:对 IoU、Params、FLOPs、FPS 分别采用 10 分位 - 90 分位归一化至 [0,1] 区间,消除量纲差异;其中 Params、FLOPs 取倒数后归一化(值越大表示效率越高)。
- 效率子分数(Eff):通过调和平均融合 Params、FLOPs、FPS 归一化结果,权重均等(各占 1/3)。
- 最终计算:U-Score = 2 ×(IoU 归一化值 × Eff)/(IoU 归一化值 + Eff),实现精度与效率的均衡量化。
我们报告了所有模型的 IoU、U-Score,以及对应的参数量、计算量和推理速度,结果如图 3 和图 1 (F) 所示。计算成本较低的模型,其 IoU 性能差异较大;而计算量更大的模型往往能实现更高精度,但需以更多资源消耗为代价。作为基线的 U-Net 在计算需求上处于中等水平,却能提供相当优异的性能。
尽管部分模型的分割精度超过 U-Net,但它们的计算开销差异显著,这使得与基线的直接对比难以进行。然而,通过 U-Score 可以发现,U-Net 在精度 - 效率的权衡上表现平平,而其他模型则呈现出更鲜明、更具区分度的结果,能够清晰划分出在权衡关系上表现优劣的方案。值得注意的是,先进模型相对 U-Net 的 IoU 提升十分有限,表明该指标已接近饱和点;而排名第一的模型与 U-Net 在 U-Score 上的巨大差距则凸显,仅以 IoU 作为评估标准已不再是医学分割任务的核心瓶颈。这些发现表明,效率正成为模型研发与实际部署中日益关键的因素。
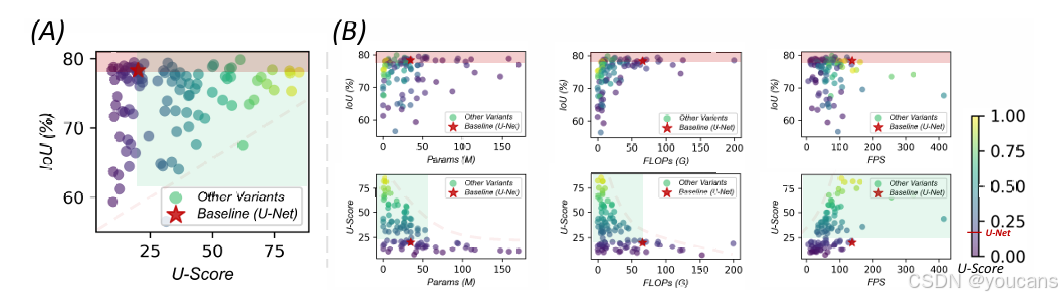
图3:IoU与U-Score的比较。红色矩形表示在IoU上表现优于U-Net的模型,绿色矩形表示在U-Score上表现优于U-Net的模型。
(A) 在100种变体中,很少有方法在IoU上优于基线U-Net,而超过一半的方法在U-Score上表现更好。
(B) 性能(IoU)与计算资源增加(FLOPs、参数、FPS)之间的关系较为复杂,而U-Score提供了一个清晰的分布,能够有效区分有利和不利的精度与效率权衡。
3. U-Bench 结果与讨论
本节将从精度、效率和泛化性多个维度呈现 U-Bench 基准测试的结果。
具体组织如下:3.1 节将对涵盖不同架构、不同发表年份的 100 种变体进行回顾性分析,探讨其发展趋势与统计发现;3.2 节将影响因素拆解为数据集和架构两方面,分析这些因素对模型性能的作用机制;3.3 节提出基于排序的顾问智能体,为研究者提供基于数据集特征和资源约束的最优模型选择方案。
3.1 过去十年的回顾性分析
发现 1:域内最优(Top-1)模型的分割精度提升有限,零样本泛化性能提升更为显著我们分析了不同架构、不同发表年份的 100 种变体(详细列表见附录 B 图 9),并报告了每年性能最优的模型,结果如图 4 所示。过去十年间,70% 的成像模态在源域和目标域的分割精度(以 IoU 衡量)均呈现稳步提升,但 IoU 提升幅度有限(平均仅 1%-2%)且表现不一致。部分模态(如光学相干断层扫描(OCT)、细胞核(Nuclei)、眼底摄影(Fundus))甚至出现停滞迹象。相比之下,80% 模态的零样本泛化性能提升更为明显,平均提升超 3%。
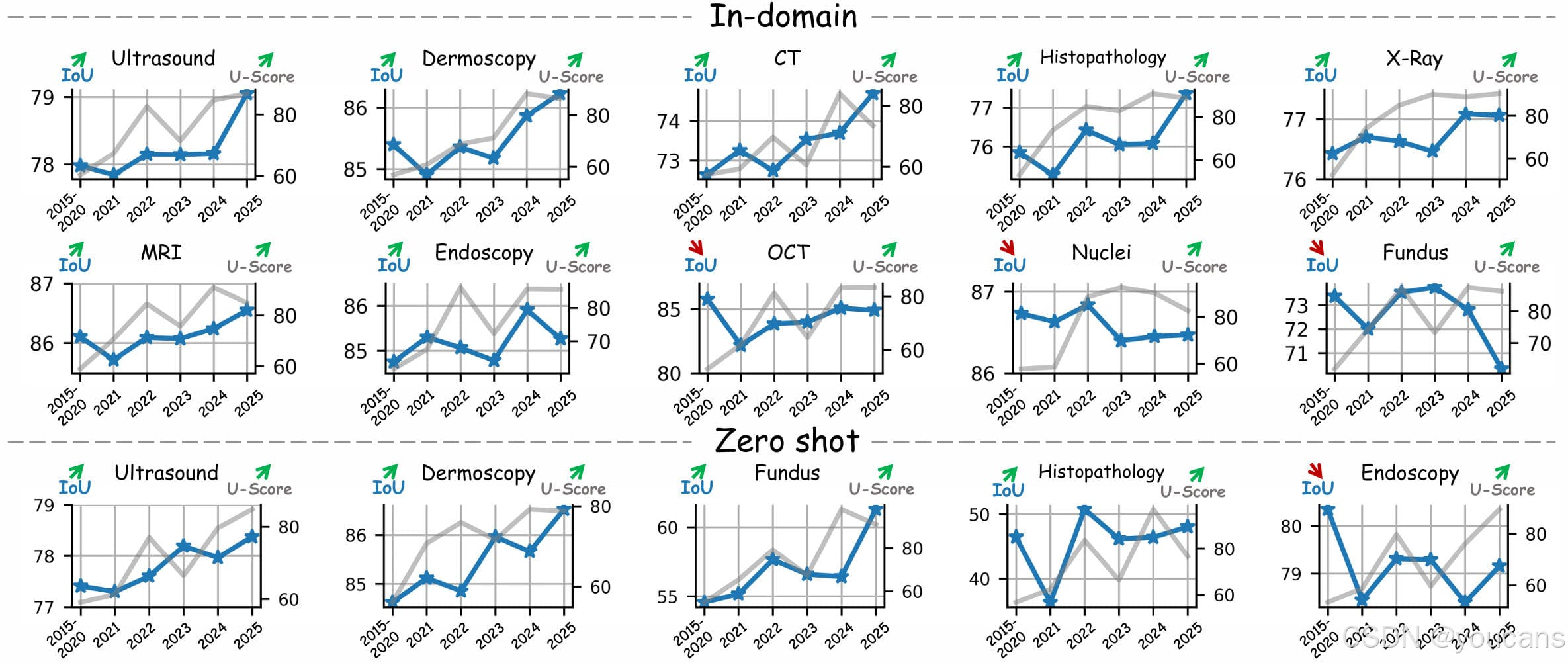
图 4:过去十年间最先进模型的性能趋势。
发现 2:尽管域内平均性能有所提升,但极少达到统计显著性;而零样本平均提升则持续具有统计显著性为严格区分 “真实性能提升” 与 “数值波动”,我们对每个变体与基线 U-Net 进行配对样本 t 检验,结果如图 1 (E) 和图 5 所示。研究发现,超过 80% 的变体未能实现统计上的显著性能提升。即便是研究最为广泛的模态(如超声、内镜、皮肤镜、计算机断层扫描(CT)、磁共振成像(MRI)),其性能提升大多也较为有限且缺乏统计显著性。仅有少数数据集(如 BUSI、TNSCUI、Kvasir、ISIC2018、Convidquex)存在持续表现更优的变体集群。与之相反,在零样本迁移实验中,对于性能优于 U-Net 的变体,75% 的模态中超过 50% 的变体均表现出统计显著性提升。
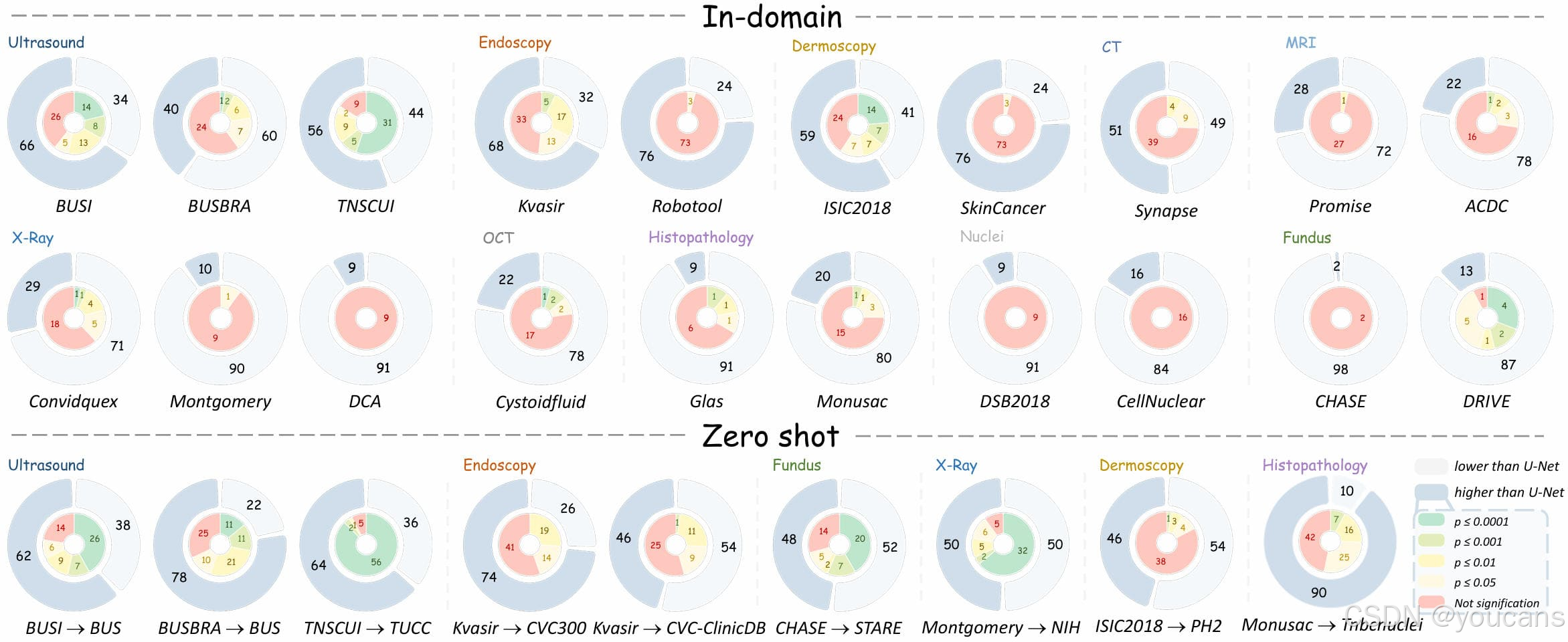
图 5:在10种模态的28个数据集上与U-Net进行统计显著性分析。
发现 1 与发现 2 的可能解释我们对上述现象给出如下可能解释:在域内评估中,具有统计显著性的性能提升通常与病灶定位任务相关,这类任务需要全局语义对比。具体而言,病灶与周围正常组织往往存在显著差异,需借助全局上下文信息建模这些差异(Zhou 等,2017; Isensee 等,2021)。近年来,随着长程依赖建模技术(如基于注意力机制的 Transformer、Mamba 等状态空间模型、RWKV 等受循环神经网络启发的混合架构)的广泛应用,架构创新日益聚焦于捕捉长程依赖关系,使得这类病灶分割任务的性能得到更为显著且稳定的提升。另一方面,长程建模技术已被证实具有更强的泛化能力(Jiang 等,2024a; Harun 等,2024; Hou 等,2025; Gu 等,2024),进而推动了零样本泛化性能的提升。相比之下,以重复性局部模式为主的模态(如细胞核、眼底摄影)从全局建模中获益有限,仅呈现出边际性提升,这也凸显了局部化机制对实现精准边界分割的补充价值。
发现 3:对效率的关注度日益提升尽管 IoU 提升有限,但 U-Score 的提升更为显著,平均增幅达 33%。这一趋势印证了我们在 2.3 节中的观点:IoU 已接近饱和点,其区分优劣方法的能力有限,表明单纯的精度已不再是分割任务的瓶颈。U-Score 的上升趋势反映了医学科研界对高效模型的重视程度不断提高,且这类模型仍有较大改进空间,也呼应了临床部署对模型的实际需求 —— 而非仅局限于实验室研究层面。
3.2 影响因素分析:架构与数据特征
3.2.1 架构因素
为分析架构选择对性能的影响,我们将 100 种模型划分为五类:CNN(卷积神经网络)、Transformer(Transformer 网络)、Mamba(Mamba 网络)、RWKV(RWKV 网络)和混合架构(Hybrid),详细说明见附录 D。我们呈现了域内和零样本场景下,按 IoU 和 U-Score 排名的全数据集 Top-10 变体(如表 2 所示),并计算了每个架构家族的平均性能(如图 6 所示)。
从分割性能(IoU)来看,混合架构通过融合局部先验信息与全局注意力机制,实现了最高精度。
如表 2(左)所示,域内和零样本场景的 Top-10 模型中,各有 5 个属于混合架构,凸显了其巨大潜力。平均而言,混合架构家族的域内性能始终最优,零样本泛化能力也极具竞争力(图 6 左),尤其在超声、内镜等以病灶为核心的任务中表现突出。新提出的 RWKV 架构家族在域内和零样本 IoU 评估中均排名第一,尽管相关研究较少,但展现出良好的发展潜力。相比之下,Mamba 架构家族的分割性能较弱,这可能与其架构设计有关 —— 尽管该架构在部分任务中具有优势,但在捕捉分割任务所需的细粒度细节或处理复杂模式时可能存在困难。
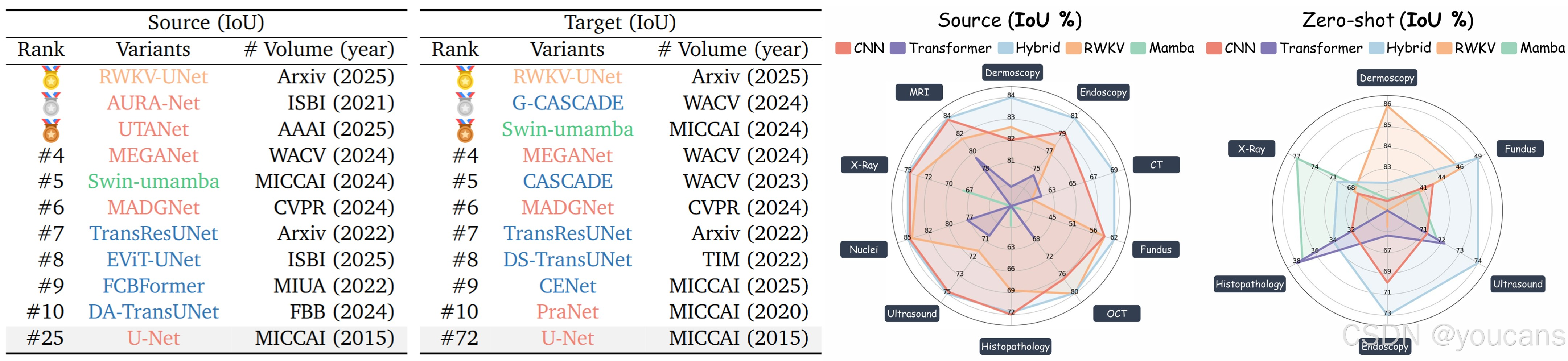
排名前10的变体:按性能(IoU)在域内和零样本设置下进行排序。
若考虑计算需求(如表 2 右所示),基于 U-Score 的排名会发生显著变化:CNN 架构家族表现领先,在域内 / 零样本场景的 Top-10 模型中分别占 7 个和 5 个。新提出的 RWKV 架构家族取得了最优的域内平均结果,零样本性能也极具竞争力(图 6 右),进一步印证了其结构优越性与潜力。相比之下,包括 Transformer 和混合架构在内的低效长程建模方法,计算需求较高,因此在 U-Score 评估中表现下降。尽管 Mamba 架构在效率方面表现出色,但精度的不稳定性削弱了其 U-Score,抵消了其效率优势。
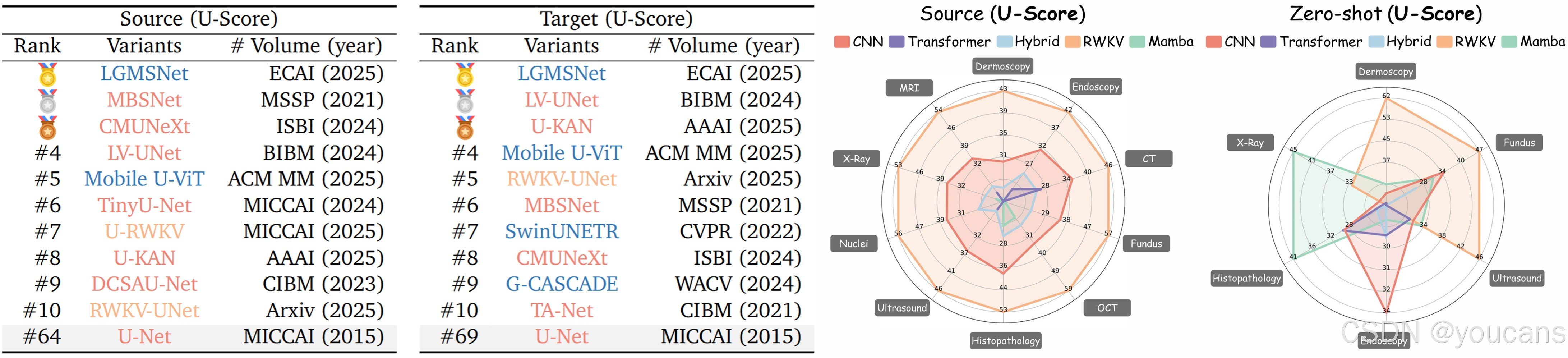
排名前10的变体:按效率(U-Score)在域内和零样本设置下进行排序。
3.2.2 数据特征因素
我们进一步从三个维度探究前景特征差异对性能的影响:前景尺度、边界清晰度和形状复杂度。附录 F.1 详细定义了目标区域、边缘和形状规则性的不同尺度划分标准。
图 7 (A) 总结了挑战性案例的特征:模糊边界是主要影响因素,常导致分割性能大幅下降,而小目标尺寸和不规则形状会进一步加剧任务难度。当这些前景属性在不同数据集间发生变化时,不同模型的性能表现也会呈现差异。如图 7 (B) 所示,与前文发现一致,混合架构在简单和复杂场景中均占据主导地位,这表明局部与全局融合机制使其能够更好地适应多样的前景特征,尤其在处理模糊边界时表现突出。基于 RWKV 的模型在捕捉不规则但轮廓清晰的形状方面展现出独特优势,反映了其建模长程轮廓的能力。尽管如此,边界模糊性以及小目标、不规则目标仍是核心挑战 —— 鉴于这类情况在医学图像中普遍存在,需要设计具有不确定性感知的模型。由于架构优势依赖于数据集特征,这些发现也凸显了 “任务感知型推荐机制” 的重要性,该机制能够实现模型与数据集属性的精准匹配。
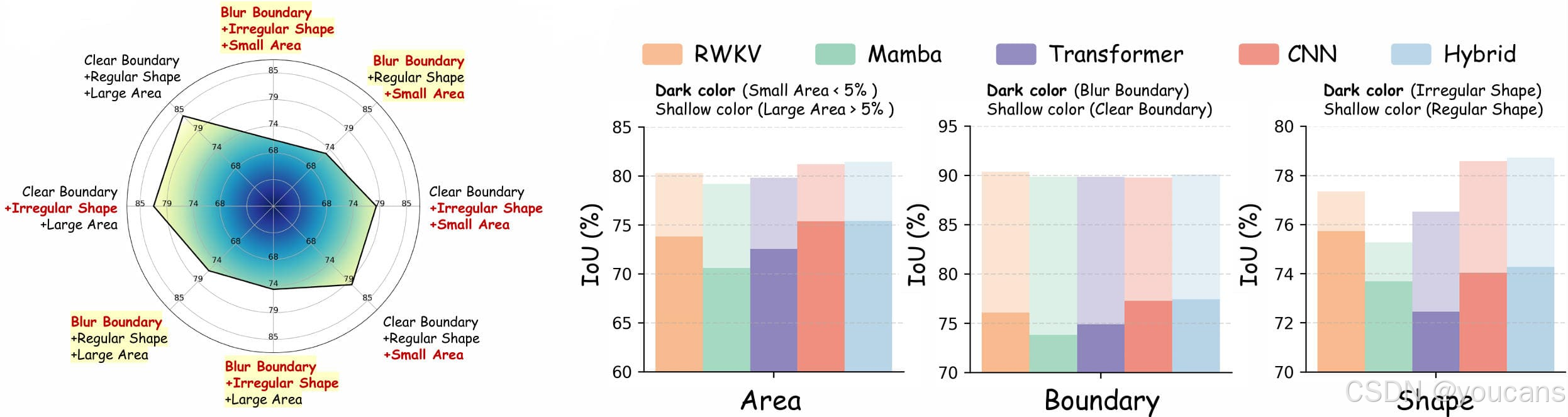
在不同前景属性下的性能分析。
3.3 模型建议智能体
基于上述分析,我们引入一种基于排序的模型顾问智能体,旨在帮助科研社区根据数据集特征和任务需求选择最合适的模型。该工具不仅能简化模型选择流程,还能协助用户权衡性能与效率,确保决策更具针对性、更贴合具体任务需求。
系统整体框架如图 8 所示。我们的顾问智能体系统利用数据集层面的特征(如成像模态、边界清晰度、形状复杂度、前景尺度)以及资源约束(存储、计算、速度),预测各类 U 型架构的适配性。该框架不再依赖人工试错方法,而是采用 XGBoost(Chen 和 Guestrin, 2016)作为推荐核心,输出最符合指定需求的候选模型及架构。关键在于,系统输出并非单一 “最优” 模型,而是一份优先级排序列表,为使用者提供更灵活的选择空间。关于推荐机制设置、数据集构建、实现细节及评估指标的更多信息,详见附录 F.2 和 F.3。
我们设计了一系列实验,验证医学图像分割领域自动模型推荐的可行性。实验采用 18 个域内数据集进行训练,预留 2 个数据集用于验证;评估指标选用归一化折损累积增益(NDCG)、平均精度均值(MAP)和斯皮尔曼相关系数(详见附录 F.3)。如表 3 所示,实验结果表明,所提出的模型顾问智能体能够有效还原与基准测试中真实 IoU 及 U-Score 排名一致的排序结果。这一结果验证了该顾问智能体系统可根据不同任务需求优先推荐适配模型,是模型选择与部署过程中可靠的辅助工具。
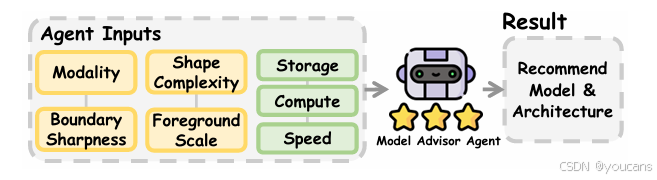
图8:我们的模型建议智能体。
4. 结论
医学图像分割领域仍面临一项核心挑战:如何对数量庞大的 U 型变体进行公平且全面的比较?为解决这一问题,我们提出 U-Bench 基准测试框架 —— 该框架通过提供全面、统计严谨且面向效率的评估方案,填补了以往评估中的关键空白。
我们的研究结果对该领域的普遍认知提出了挑战:尽管许多变体表现出性能提升,但在域内评估中极少能达到统计显著性;与之相反,零样本泛化性能取得了显著进步,凸显了模型跨域泛化的巨大潜力。此外,新提出的 U-Score 指标兼顾性能与效率,标志着研究范式正从 “单纯追求精度” 向 “平衡性能与效率” 转变。
基于对模型架构和数据集特征的分析见解,我们设计了一种基于排序的模型顾问智能体,将大规模评估结果转化为切实可行的指导,帮助研究者选择适配特定任务的模型。通过将 U-Bench 作为开源平台发布,我们为科研社区提供了一个稳健、可复现的工具,以推动分割领域的研究进展,助力开发兼具高精度与临床部署可行性的模型。
5. 附录B:U-Net 的各类变体
附录 B 将全面概述 U-Net 网络的各类变体,包括网络架构设计及现有医学分割基准测试平台。
5.1 模型架构
作为医学图像分割的核心架构,近年来在特征表示能力提升、长程依赖建模技术发展以及效率与精度权衡需求的推动下,U-Net 已演化出众多变体。本节将基于核心范式与设计动机,对这些 U-Net 变体进行分类梳理,系统追溯其从基础构建到融合创新的演化路径。图 9 总结了 U-Net 变体随时间的发展历程。
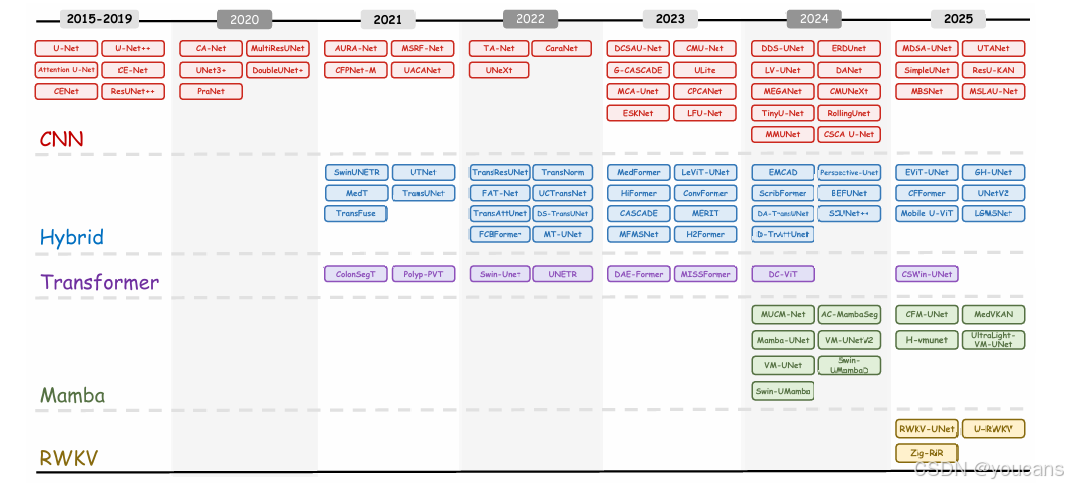
图9:所有评估模型的时间和架构分布.
-
卷积神经网络(CNN)主导的 U 型网络(2015-2021 年:基础奠定阶段)
这类 U 型网络以卷积神经网络为唯一骨干网络,通过卷积操作提取局部特征,并利用固定跳接连接融合多尺度信息,为医学图像分割领域的 “编码器 - 解码器” 范式奠定了基础。其优势在于能精准捕捉局部细节(如纹理、边缘),架构相对轻量化,训练过程稳定,为后续复杂变体提供了坚实的设计基准。但卷积操作的局部感受野限制了全局语义信息的建模能力,且固定跳接连接易导致编码器与解码器间存在语义鸿沟,进而制约性能提升。 -
Transformer 驱动的 U 型网络(2021-2023 年:范式转型阶段)
该类变体引入 Transformer 架构(包括视觉 Transformer(Dosovitskiy 等,2020)、Swin Transformer(Liu 等,2021)等衍生模型),替代或增强传统 CNN 骨干网络,借助自注意力机制有效建模长程依赖关系。然而,自注意力机制的计算复杂度随序列长度呈二次增长,导致推理效率较低;同时,这类模型对小规模医学数据集的适应性较差,易因数据不足发生过拟合,难以满足临床边缘设备严苛的实时性需求。 -
基于状态空间模型(SSM)与循环范式的 U 型网络(2023-2025 年:效率导向阶段)
近期研究致力于用线性时间复杂度的替代方案取代二次复杂度的自注意力机制。一类研究采用状态空间模型(如 Mamba(Gu 和 Dao, 2023)),通过选择性状态更新捕捉长程依赖,同时实现线性复杂度,显著提升了推理效率与小样本适应性;另一类互补研究引入 RWKV(Peng 等,2023)—— 一种受循环神经网络启发的模型,融合了 Transformer 的表达能力与 RNN 的循环特性,能够高效处理序列数据,并在不同输入长度下具备更强泛化性。这两类范式共同缓解了 Transformer 在计算成本与数据依赖性上的局限性。 -
多范式融合 U 型网络(混合网络)(2020-2025 年:融合创新阶段)
该阶段旨在整合 CNN 的局部特征提取优势、Transformer 的全局语义建模能力、状态空间模型与循环范式的效率优势。通过融合不同架构,实现精度、效率与泛化性的平衡。这类网络变体能够适配多模态成像、跨中心数据异质性等复杂临床场景,显著提升分割结果的实用价值。但随之而来的是架构设计复杂度大幅增加,不同范式模块间的协调机制(如特征交互时机、权重分配)仍需进一步优化。
上述四类 U 型网络变体的发展,遵循 “局部细化→全局关联→效率考量→多范式协同” 的技术演化路径,反映了临床需求从静态、单一场景分割,向更高效、更具泛化性、适配多样化条件的解决方案转变的趋势。
5.2 医学分割基准测试平台
为填补 U-Net 系统评估领域的研究空白,我们将以往的分割评估基准测试平台与本文提出的 U-Bench 进行全面对比,明确 U-Bench 的创新定位。
B.2.1 相关研究
在深度学习架构与大规模数据集的推动下,医学图像分割领域取得了快速发展。然而,由于评估协议不一致、数据集多样性有限以及对部署约束的考量不足,许多已报道的进展在有效性和可复现性方面受到了质疑。
- TorchStone(Bassi 等,2024)通过引入大规模腹部器官分割协同基准测试平台,解决了部分上述局限性。该平台利用了来自全球多家医院的多样化计算机断层扫描(CT)数据,虽强调了分布外泛化能力评估与标准化评估流程,但其应用范围仅限于单一解剖区域和模态,难以用于评估模型更广泛的架构能力。
- MedSegBench(Zhou 等,2025)扩展了模态覆盖范围,整合了超声、磁共振成像(MRI)、X 射线等 35 个数据集,提供了标准化的数据划分,并对多种编码器 - 解码器变体进行了评估,旨在推动通用分割模型的研发。但该平台仍聚焦于少量架构,缺乏对模型稳健性、效率及跨范式对比的全面分析。
- nnWNet(Zhou 等,2025)提出了架构改进方案,在 U-Net 框架内融合卷积与 Transformer,以满足局部和全局特征持续传递的需求。尽管该模型在多个二维(2D)和三维(3D)数据集上进行了基准测试,但其评估仅局限于少量模型,且缺乏系统性的效率分析。
- nnU-Net Revisited(Isensee 等,2024)对近期的架构相关主张进行了批判性验证,结果表明,若训练资源充足,配置合理的基于 CNN 的 U-Net 仍能达到或优于较新的基于 Transformer 和 Mamba 的模型。该研究凸显了严谨基线与计算可复现性的重要性,但未提供一个多模态、多数据集的框架来对比大量变体。
综上,这些研究均表明,业界亟需一个统一、统计严谨且全面的基准测试平台,能够跨多样模态、数据集和部署指标,对广泛的 U-Net 变体进行系统性评估。
B.2.2 U-Bench 的针对性改进
如表 1 所示,现有医学图像分割基准测试平台存在模态覆盖有限、评估维度不足、架构范围狭窄以及缺乏数据集特异性分析等问题,这些均阻碍了对模型泛化能力的全面评估。为填补这些空白,U-Bench 设计了三项针对性创新,构建了更全面且贴合临床实际的评估框架,同时契合其核心目标:在 28 个数据集和 10 种模态上评估 100 种 U-Net 变体,引入平衡性能与效率的 U-Score,并实现公平、可复现的基准测试。
-
多模态与全任务覆盖
U-Bench 涵盖 10 种主要医学成像模态(超声、皮肤镜、内镜、眼底摄影、组织病理学、细胞核、X 射线、磁共振成像(MRI)、计算机断层扫描(CT)、光学相干断层扫描(OCT)),整合了 28 个数据集(样本量范围:20-17,000 例)。其覆盖的任务从宏观器官分割(如肺部 CT、心脏 MRI 分割)到微观结构分割(如组织病理学细胞核、视网膜微血管分割),并采用标准化的训练 / 测试集划分。该设计可测试模型的跨模态适配能力,与真实临床多模态诊断流程保持一致。 -
多维度评估体系除传统精度指标(IoU、Dice 相似系数)外,U-Bench 新增三个关键评估维度,并通过统一的 U-Score 量化临床实用价值:
- 计算效率:标准化报告模型参数量(单位:M)、推理计算量(单位:G)和推理速度(FPS),反映模型在资源受限设备上的部署可行性;
- 泛化性能:在 8 个未见过的目标数据集(与 20 个训练源数据集不同)上进行零样本迁移测试,评估模型对域转移(如跨中心超声数据、未见过的皮肤镜病灶)的稳健性;
- 统计显著性:通过每个变体与原始 U-Net 的配对 t 检验(p < 0.05 为显著),验证性能提升的可靠性;
- U-Score:采用分位数归一化与加权调和平均的综合指标,平衡精度与效率,搭建学术性能与临床部署价值之间的桥梁。
-
大规模可复现验证
U-Bench 包含 100 种公开可用的 U-Net 变体,覆盖 2015-2025 年的主流架构(CNN、Transformer、Mamba、RWKV、混合设计)。为确保可复现性,所有模型均采用官方实现、预训练权重(如有)及深度监督策略(如适用)。
5.3 实验评估的数据集
我们在表4中总结了本文中使用的数据集统计信息。
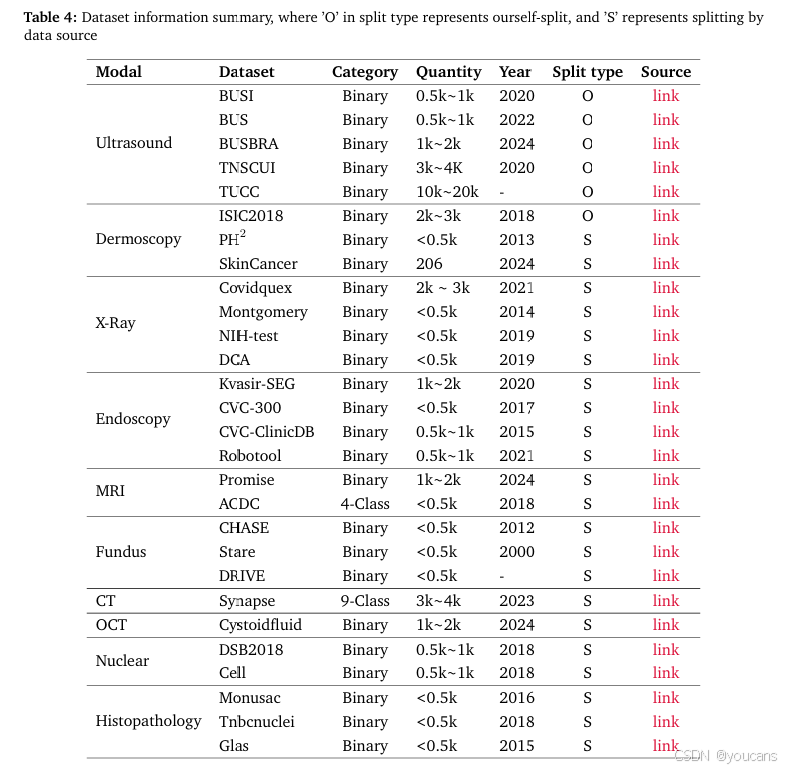
该表详细列出了用于实验评估的数据集,涵盖了10种核心成像模态,包括超声(例如BUSI)、皮肤镜(例如ISIC2018)、内窥镜(例如Kvasir-SEG)、眼底(例如CHASE)、组织病理学(例如Glas)、核医学(例如DSB2018)、X光(例如Montgomery)、MRI(例如ACDC)、CT(例如Synapse)和OCT(例如Cystoidfluid)。
对于每个数据集,我们提供了关键信息,例如分割类别(二分类或多分类)、样本数量、发布年份和基本描述。所有使用到的数据集均为公开可用,因此我们在相关参考文献和补充表格中提供了访问链接。详细信息可在表4中找到。以下是数据集的简要描述:
- BUSI.乳腺超声图像(BUSI)数据集(Al-Dhabyani等人,2020年),于2018年从600名女性患者中收集,包含133例正常、487例良性以及210例恶性病例及其对应的真值标签。数据标签是通过超声扫描检查乳腺癌病变区域获得的。
- BUS.乳腺超声(BUS)公开数据集(Zhang等人,2022年)包含562张图像(306例良性,256例恶性),通过五台超声设备收集,用于泛化实验。数据标签是通过超声扫描检查乳腺癌病变(或非病变)区域获得的。
- BUSBRA.BUS-BRA数据集(Gómez-Flores等人,2024年)包含1875张匿名图像,来自1064名患者(对应722例良性病例和342例恶性病例),通过四台超声扫描仪获取。数据标签是通过超声扫描检查乳腺癌病变(或非病变)区域获得的。
- TNSCUI.甲状腺结节超声图像分割与分类2020数据集1包含来自中国甲状腺和乳腺超声人工智能联盟的3644个病例。数据标签是通过甲状腺超声检查获得的甲状腺结节区域。
- TUCC.甲状腺超声(TUCC)数据集2从167名患者中收集数据,包括192个活检确认的结节。数据标签是通过甲状腺超声检查获得的甲状腺结节区域。
- ISIC2018.ISIC2018数据集(Codella等人,2018年)是一个用于病变分割的大规模皮肤镜数据集,包含2594张皮肤病变图像。数据标签是通过皮肤镜成像获得的皮肤疾病中的黑色素瘤(或非病变)区域。
- PH2.PH2数据库(Mendonça等人,2013年)包含200张带有手动分割和临床诊断的皮肤镜图像。数据标签是通过皮肤镜成像获得的皮肤疾病中的黑色素瘤(或非病变)区域。
- SkinCancer.SkinCancer数据集(Kuş和Aydin,2024年)包含从DermIS和DermQuest提取的206张皮肤镜样本。数据标签是通过皮肤镜成像获得的皮肤疾病中的黑色素瘤(或非病变)区域。
- Covidquex.Covidquex数据集(Kuş和Aydin,2024年)包含2913张胸部X光图像(256×256像素),用于二元分割。该数据集标记了胸部X光上的COVID感染区域。
- Montgomery.Montgomery数据集(Jaeger等人,2014年)包含138张胸部X光图像(80张正常,58张带有肺结核)。数据标签是肺部X光上的肺结核病变(或非病变)区域。
- NIH-test.NIH-test数据集(Tang等人,2019年)是一个手动标注的胸部X光数据集,包含100张肺部掩膜。数据标签是胸部X光上的肺部分割。
- DCA.DCA数据集(Kuş和Aydin,2024年)包含134张眼底图像(300×300像素)。数据标签是眼底图像的血管分割。
- Kvasir.Kvasir数据集(Jha等人,2020b)包含1000张胃肠息肉图像及其对应的真值。数据标签是胃肠内窥镜成像的病理区域。
- CVC-300.CVC-300数据集(Vázquez等人,2017年)包含60张结肠镜息肉图像(500×574像素)。数据标签是胃肠内窥镜成像的病理区域。
- CVC-ClinicDB.CVC-ClinicDB数据集(Bernal等人,2015年)包含来自29个结肠镜序列的612张图像。数据标签是胃肠内窥镜成像的病理区域。
- Robotool.Robotool数据集(Kuş和Aydin,2024年)包含从多个手术视频中提取的500张图像。数据标签是内窥镜成像的器械区域。
- Promise.Promise数据集(Kuş和Aydin,2024年)包含1473张前列腺MRI样本(512×512像素)。
- ACDC.ACDC数据集(Bernard等人,2018年)包含100张心脏MRI扫描图像。数据标签是心脏分割中的左心室(LV)、右心室(RV)和心肌(MYO)。
- CHASE.CHASE数据集(Fraz等人,2012年)包含28张视网膜图像(每只眼睛1张,共14名儿童)。数据标签是眼底图像的血管区域。
- Stare.Stare数据集(Hoover等人,2000年)包含20张带有手动标注的视网膜血管图像。数据标签是眼底图像的血管区域。
- DRIVE.DRIVE数据集(Staal等人,2004年)是从荷兰糖尿病视网膜病变筛查项目中收集的。数据标签是眼底图像的血管区域。
- Cell.Cell数据集(Kuş和Aydin,2024年)包含670张分辨率为320×256像素的细胞核图像。数据标签是细胞核分割区域。
- Glas.Glas数据集(Sirinukunwattana等人,2015年)包含165张用于腺体分割的H&E染色切片图像。数据标签是H&E图像中的腺体病变(或非病变)区域。
- Monusac.Monusac数据集(Kuş和Aydin,2024年)包含310张H&E染色数字组织图像。数据标签是H&E染色组织学图像中的细胞核区域。
- Tnbcnuclei.Tnbcnuclei数据集(Kuş和Aydin,2024年)包含50张用于二元分割的病理样本。数据标签是H&E染色组织学图像中的细胞核区域。
- Synapse.Synapse多器官数据集包含30张带有8器官分割的腹部CT扫描图像。数据标签是8个腹部器官(主动脉、胆囊、左肾、右肾、肝脏、胰腺、脾脏、胃)。
- Cystoidfluid.Cystoidfluid数据集(Kuş和Aydin,2024年)包含1006张光学相干断层扫描图像。数据集标记了视网膜的囊样黄斑水肿(CME)区域。
- DSB2018.DSB2018数据集(Hamilton,2018年)包含670张H&E染色核图像。数据标签是细胞核分割区域。
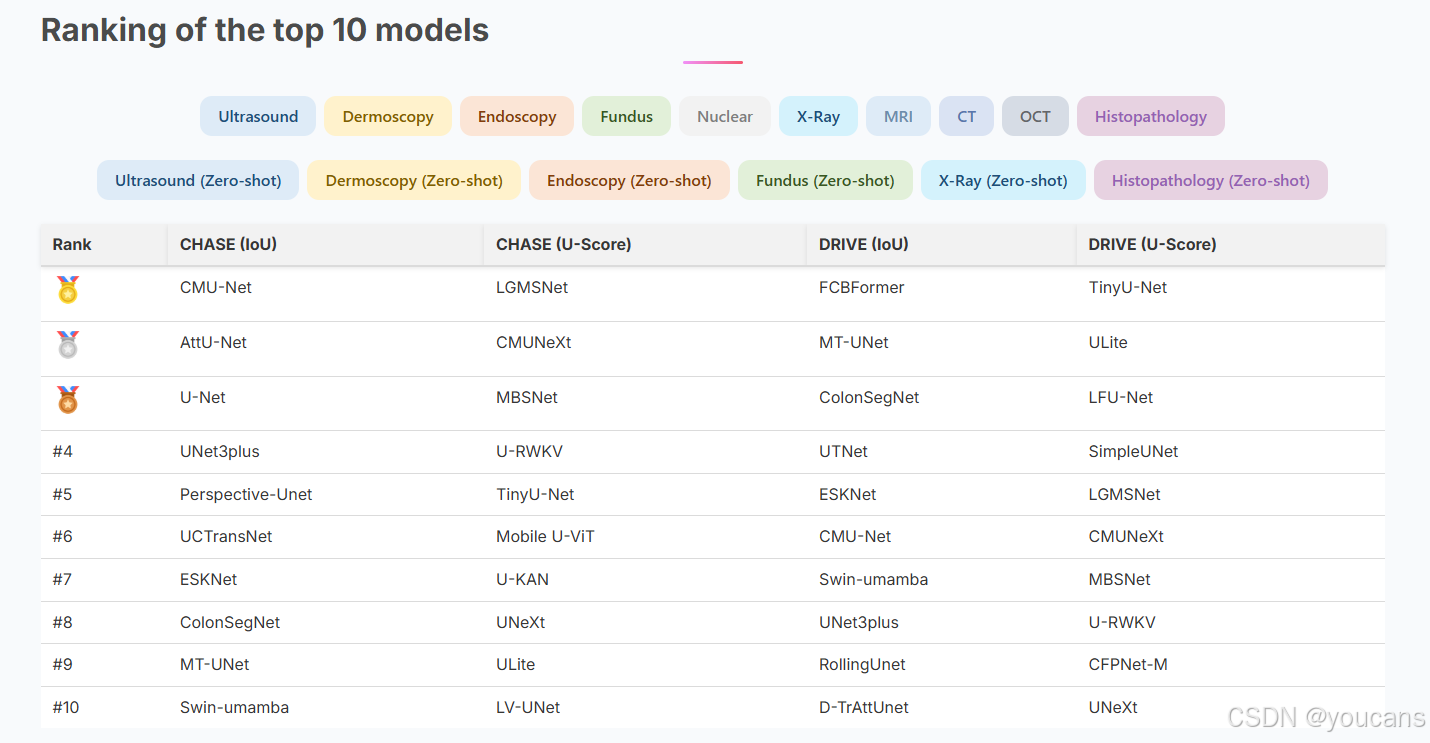
5.4 视网膜血管分割 Top10 模型
import os
cpu_num = 1
os.environ['OMP_NUM_THREADS'] = str(cpu_num)
os.environ['OPENBLAS_NUM_THREADS'] = str(cpu_num)
os.environ['MKL_NUM_THREADS'] = str(cpu_num)
os.environ['VECLIB_MAXIMUM_THREADS'] = str(cpu_num)
os.environ['NUMEXPR_NUM_THREADS'] = str(cpu_num)
import random
import argparse
from tqdm import tqdm
import matplotlib.pyplot as plt
import json
import logging
import numpy as np
import torch
torch.set_num_threads(cpu_num)
torch.multiprocessing.set_sharing_strategy('file_system')
import wandb
import torch.optim as optim
from monai.losses import DiceCELoss, DiceLoss
import csv
device = torch.device(f'cuda:0' if torch.cuda.is_available() else 'cpu')
print(f"Using device: {device}")
from models import build_model
import utils.losses as losses
from utils.metrics_medpy import get_metrics
from utils.util import AverageMeter
import tempfile
from torch.utils.tensorboard import SummaryWriter
from dataloader.dataloader import getDataloader,getZeroShotDataloader
import torch.nn.functional as F
parser = argparse.ArgumentParser()
parser.add_argument('--gpu', type=str, default="7", help='gpu')
temp_args, _ = parser.parse_known_args()
os.environ["CUDA_VISIBLE_DEVICES"] = temp_args.gpu
print(f"Set CUDA_VISIBLE_DEVICES to {os.environ['CUDA_VISIBLE_DEVICES']}")
def convert_to_numpy(data):
if isinstance(data, torch.Tensor):
return data.cpu().numpy()
elif isinstance(data, dict):
return {key: convert_to_numpy(value) for key, value in data.items()}
elif isinstance(data, list):
return [convert_to_numpy(item) for item in data]
else:
return data
def seed_torch(seed):
torch.manual_seed(seed)
torch.cuda.manual_seed(seed)
torch.cuda.manual_seed_all(seed)
np.random.seed(seed)
random.seed(seed)
torch.backends.cudnn.deterministic = True
torch.backends.cudnn.benchmark = False
os.environ["PYTHONHASHSEED"] = str(seed)
torch.use_deterministic_algorithms(True,warn_only=True)
os.environ["CUBLAS_WORKSPACE_CONFIG"] = ":4096:8"
def parse_arguments():
parser = argparse.ArgumentParser()
parser.add_argument('--model', type=str, default="U_Net", help='model')
parser.add_argument('--base_dir', type=str, default="./data/busi", help='data base dir')
parser.add_argument('--dataset_name', type=str, default="busi", help='dataset_name')
parser.add_argument('--train_file_dir', type=str, default="train.txt", help='train_file_dir')
parser.add_argument('--val_file_dir', type=str, default="val.txt", help='val_file_dir')
parser.add_argument('--base_lr', type=float, default=0.01,
help='segmentation network learning rate')
parser.add_argument('--batch_size', type=int, default=8,
help='batch_size per gpu')
parser.add_argument('--gpu', type=str, default="7", help='gpu')
parser.add_argument('--max_epochs', type=int, default=2, help='epoch')
parser.add_argument('--seed', type=int, default=41, help='seed')
parser.add_argument('--img_size', type=int, default=256, help='img_size')
parser.add_argument('--num_classes', type=int, default=1, help='img_size')
parser.add_argument('--input_channel', type=int, default=3, help='img_size')
parser.add_argument('--resume', action='store_true', help='Resume training from checkpoint')
parser.add_argument('--exp_name', type=str, default="default_exp", help='Experiment name')
parser.add_argument('--zero_shot_base_dir', type=str, default="", help='zero_base_dir')
parser.add_argument('--zero_shot_dataset_name', type=str, default="", help='zero_shot_dataset_name')
parser.add_argument('--do_deeps', type=bool, default=False, help='Use deep supervision')
parser.add_argument('--model_id', type=int, default=0, help='model_id')
parser.add_argument('--just_for_test', type=bool, default=0, help='just for test')
parser.add_argument('--just_for_zero_shot', type=bool, default=0, help='just for test')
args = parser.parse_args()
seed_torch(args.seed)
return args
args = parse_arguments()
def deep_supervision_loss(outputs, label_batch, loss_metric,weights=None):
num=len(outputs)
total_loss = 0.0
for i, output in enumerate(outputs):
if output.shape[1:] != label_batch.shape[1:]:
output = F.interpolate(output, size=label_batch.shape[1:], mode='bilinear', align_corners=True)
loss = loss_metric(output, label_batch)
total_loss += loss
return total_loss/ num
def load_model(args, model_best_or_final="best"):
exp_save_dir= args.exp_save_dir
model = build_model(args, input_channel=args.input_channel, num_classes=args.num_classes).to(device)
if model_best_or_final == "best":
model_path = os.path.join(exp_save_dir, f'checkpoint_best.pth')
else:
model_path = os.path.join(exp_save_dir, f'checkpoint_final.pth')
checkpoint = torch.load(model_path, map_location=device, weights_only=False)
if 'state_dict' in checkpoint:
state_dict = checkpoint['state_dict']
else:
state_dict = checkpoint
model.load_state_dict(state_dict)
model.to(device)
return model, model_path
def zero_shot(args,logger,model=None,wandb=None):
valloader = getZeroShotDataloader(args)
if model is None:
model,model_path = load_model(args)
logger.info("train file dir:{} val file dir:{}".format(args.train_file_dir, args.val_file_dir))
criterion = losses.__dict__['BCEDiceLoss']().to(device)
avg_meters = {'loss': AverageMeter(),
'iou': AverageMeter(),
'val_loss': AverageMeter(),
'val_iou': AverageMeter(),
'SE': AverageMeter(),
'PC': AverageMeter(),
'F1': AverageMeter(),
'ACC': AverageMeter()
}
model.eval()
with torch.no_grad():
for i_batch, sampled_batch in tqdm(enumerate(valloader), total=len(valloader), desc="Zero-shot Validation"):
input, target = sampled_batch['image'], sampled_batch['label']
input = input.to(device)
target = target.to(device)
output = model(input)
output = output[-1] if args.do_deeps else output
loss = criterion(output, target)
iou, _, SE, PC, F1, _, ACC = get_metrics(output, target)
avg_meters['val_loss'].update(loss.item(), input.size(0))
avg_meters['val_iou'].update(iou, input.size(0))
avg_meters['SE'].update(SE, input.size(0))
avg_meters['PC'].update(PC, input.size(0))
avg_meters['F1'].update(F1, input.size(0))
avg_meters['ACC'].update(ACC, input.size(0))
logger.info(f"zero shot on {args.zero_shot_dataset_name}")
logger.info('val_loss %.4f - val_iou %.4f - val_SE %.4f - val_PC %.4f - val_F1 %.4f - val_ACC %.4f'
% (avg_meters['val_loss'].avg, avg_meters['val_iou'].avg, avg_meters['SE'].avg,
avg_meters['PC'].avg, avg_meters['F1'].avg, avg_meters['ACC'].avg))
zero_shot_result = {"zeroshot_loss":avg_meters['val_loss'].avg, "zeroshot_iou":avg_meters['val_iou'].avg, "zeroshot_SE":avg_meters['SE'].avg,
"zeroshot_PC":avg_meters['PC'].avg, "zeroshot_F1":avg_meters['F1'].avg, "zeroshot_ACC":avg_meters['ACC'].avg}
zero_shot_result = convert_to_numpy(zero_shot_result)
return zero_shot_result
def init_dir(args):
exp_save_dir = f'./output/{args.model}/{args.dataset_name}/{args.exp_name}/'
os.makedirs(exp_save_dir, exist_ok=True)
args.exp_save_dir = exp_save_dir
config_file_path = os.path.join(exp_save_dir, f'config.json')
args_dict = vars(args)
with open(config_file_path, 'w') as f:
json.dump(args_dict, f, indent=4)
print(f"Config saved to {config_file_path}")
writer = SummaryWriter(log_dir=f'{exp_save_dir}/tensorboard_logs/')
log_file = os.path.join(exp_save_dir, f'training.log')
logger = logging.getLogger(__name__)
logger.setLevel(logging.INFO)
formatter = logging.Formatter('%(asctime)s - %(levelname)s - %(message)s')
file_handler = logging.FileHandler(log_file)
file_handler.setFormatter(formatter)
logger.addHandler(file_handler)
stream_handler = logging.StreamHandler()
stream_handler.setFormatter(formatter)
logger.addHandler(stream_handler)
model = build_model(config=args,input_channel=args.input_channel, num_classes=args.num_classes).to(device)
return exp_save_dir, writer, logger, model#, wandb
def validate(args,logger,model):
trainloader,valloader = getDataloader(args)
criterion = losses.__dict__['BCEDiceLoss']().to(device)
avg_meters = {'loss': AverageMeter(),
'iou': AverageMeter(),
'val_loss': AverageMeter(),
'val_iou': AverageMeter(),
'SE': AverageMeter(),
'PC': AverageMeter(),
'F1': AverageMeter(),
'ACC': AverageMeter()
}
model.eval()
with torch.no_grad():
for i_batch, sampled_batch in enumerate(valloader):
input, target = sampled_batch['image'], sampled_batch['label']
input = input.to(device)
target = target.to(device)
output = model(input)
output = output[-1] if args.do_deeps else output
loss = criterion(output, target)
iou, _, SE, PC, F1, _, ACC = get_metrics(output, target)
avg_meters['val_loss'].update(loss.item(), input.size(0))
avg_meters['val_iou'].update(iou, input.size(0))
avg_meters['SE'].update(SE, input.size(0))
avg_meters['PC'].update(PC, input.size(0))
avg_meters['F1'].update(F1, input.size(0))
avg_meters['ACC'].update(ACC, input.size(0))
val_metric_dict = {
"val_loss":avg_meters['val_loss'].avg, "val_iou":avg_meters['val_iou'].avg, "val_SE":avg_meters['SE'].avg,
"val_PC":avg_meters['PC'].avg, "val_F1":avg_meters['F1'].avg, "val_ACC":avg_meters['ACC'].avg
}
val_metric_dict = convert_to_numpy(val_metric_dict)
return val_metric_dict
def train(args,exp_save_dir, writer, logger, model):
start_epoch = 0
base_lr = args.base_lr
trainloader, valloader = getDataloader(args)
model_parameters = sum(p.numel() for p in model.parameters() if p.requires_grad)
logger.info(f"model:{args.model} model_parameters:{model_parameters}")
logger.info(f"train file dir:{args.train_file_dir} val file dir:{args.val_file_dir}")
logger.info(f"{len(trainloader)} iterations per epoch")
optimizer = optim.SGD(model.parameters(), lr=base_lr, momentum=0.9, weight_decay=0.0001)
criterion = losses.__dict__['BCEDiceLoss']().to(device)
train_metric_dict = {
"best_iou": 0,
"best_epoch": 0,
"best_iou_withSE": 0,
"best_iou_withPC": 0,
"best_iou_withF1": 0,
"best_iou_withACC": 0,
"last_iou": 0,
"last_SE": 0,
"last_PC": 0,
"last_F1": 0,
"last_ACC": 0
}
max_epoch = args.max_epochs
max_iterations = len(trainloader) * max_epoch
train_loss_list = []
train_iou_list = []
loss_list = []
iou_list = []
f1_list = []
if args.resume:
checkpoint_path = os.path.join(exp_save_dir, f'checkpoint.pth')
if os.path.exists(checkpoint_path):
checkpoint = torch.load(checkpoint_path)
model.load_state_dict(checkpoint['state_dict'])
optimizer.load_state_dict(checkpoint['optimizer'])
start_epoch = checkpoint['epoch']
train_metric_dict["best_iou"] = checkpoint['best_iou']
logger.info(f"Resuming training from epoch {start_epoch} with best IoU {train_metric_dict['best_iou']}")
iter_num = start_epoch * len(trainloader)
for epoch_num in tqdm(range(start_epoch, max_epoch), desc='Training Progress'):
model.train()
avg_meters = {'loss': AverageMeter(),
'iou': AverageMeter(),
'val_loss': AverageMeter(),
'val_iou': AverageMeter(),
'SE': AverageMeter(),
'PC': AverageMeter(),
'F1': AverageMeter(),
'ACC': AverageMeter()
}
for i_batch, sampled_batch in enumerate(trainloader):
volume_batch, label_batch = sampled_batch['image'], sampled_batch['label']
volume_batch, label_batch = volume_batch.to(device), label_batch.to(device)
if args.do_deeps:
outputs = model(volume_batch)
loss = deep_supervision_loss(outputs=outputs,label_batch=label_batch,loss_metric=criterion)
outputs=outputs[-1]
else:
outputs = model(volume_batch)
loss = criterion(outputs, label_batch)
iou, dice, _, _, _, _, _ = get_metrics(outputs, label_batch)
optimizer.zero_grad()
loss.backward()
optimizer.step()
lr_ = base_lr * (1.0 - iter_num / max_iterations) ** 0.9
for param_group in optimizer.param_groups:
param_group['lr'] = lr_
iter_num += 1
avg_meters['loss'].update(loss.item(), volume_batch.size(0))
avg_meters['iou'].update(iou, volume_batch.size(0))
model.eval()
with torch.no_grad():
for i_batch, sampled_batch in enumerate(valloader):
input, target = sampled_batch['image'], sampled_batch['label']
input = input.to(device)
target = target.to(device)
output = model(input)
output = output[-1] if args.do_deeps else output
loss = criterion(output, target)
iou, _, SE, PC, F1, _, ACC = get_metrics(output, target)
avg_meters['val_loss'].update(loss.item(), input.size(0))
avg_meters['val_iou'].update(iou, input.size(0))
avg_meters['SE'].update(SE, input.size(0))
avg_meters['PC'].update(PC, input.size(0))
avg_meters['F1'].update(F1, input.size(0))
avg_meters['ACC'].update(ACC, input.size(0))
train_loss_list.append(avg_meters['loss'].avg)
loss_list.append(avg_meters['val_loss'].avg)
if isinstance(avg_meters['val_iou'].avg, torch.Tensor):
iou_list.append(avg_meters['val_iou'].avg.cpu().numpy())
else:
iou_list.append(avg_meters['val_iou'].avg)
if isinstance(avg_meters['F1'].avg, torch.Tensor):
f1_list.append(avg_meters['F1'].avg.cpu().numpy())
else:
f1_list.append(avg_meters['F1'].avg)
writer.add_scalar('Train/Loss', avg_meters['loss'].avg, epoch_num)
writer.add_scalar('Train/IOU', avg_meters['iou'].avg, epoch_num)
writer.add_scalar('Val/Loss', avg_meters['val_loss'].avg, epoch_num)
writer.add_scalar('Val/IOU', avg_meters['val_iou'].avg, epoch_num)
writer.add_scalar('Val/SE', avg_meters['SE'].avg, epoch_num)
writer.add_scalar('Val/PC', avg_meters['PC'].avg, epoch_num)
writer.add_scalar('Val/F1', avg_meters['F1'].avg, epoch_num)
writer.add_scalar('Val/ACC', avg_meters['ACC'].avg, epoch_num)
log_info = (
f"epoch [{epoch_num}/{max_epoch}] train_loss: {avg_meters['loss'].avg:.4f}, train_iou: {avg_meters['iou'].avg:.4f} "
f"- val_loss {avg_meters['val_loss'].avg:.4f} - val_iou {avg_meters['val_iou'].avg:.4f} "
f"- val_SE {avg_meters['SE'].avg:.4f} - val_PC {avg_meters['PC'].avg:.4f} "
f"- val_F1 {avg_meters['F1'].avg:.4f} - val_ACC {avg_meters['ACC'].avg:.4f}"
)
logger.info(log_info)
if avg_meters['val_iou'].avg > train_metric_dict["best_iou"]:
train_metric_dict["best_iou"] = avg_meters['val_iou'].avg
train_metric_dict["best_epoch"] = epoch_num
train_metric_dict["best_iou_withSE"] = avg_meters['SE'].avg
train_metric_dict["best_iou_withPC"] = avg_meters['PC'].avg
train_metric_dict["best_iou_withF1"] = avg_meters['F1'].avg
train_metric_dict["best_iou_withACC"] = avg_meters['ACC'].avg
model_save_path = os.path.join(
exp_save_dir,
f'checkpoint_best.pth'
)
torch.save({
'state_dict': model.state_dict(),
'config': vars(args),
'epoch': epoch_num + 1,
}, model_save_path)
print("=> saved best model with config")
if epoch_num == max_epoch - 1:
train_metric_dict["last_iou"] = avg_meters['val_iou'].avg
train_metric_dict["last_SE"] = avg_meters['SE'].avg
train_metric_dict["last_PC"] = avg_meters['PC'].avg
train_metric_dict["last_F1"] = avg_meters['F1'].avg
train_metric_dict["last_ACC"] = avg_meters['ACC'].avg
checkpoint_path = os.path.join(exp_save_dir, f'checkpoint_final.pth')
torch.save({
'epoch': epoch_num + 1,
'state_dict': model.state_dict(),
'optimizer': optimizer.state_dict(),
'best_iou': train_metric_dict["best_iou"],
'config': vars(args),
}, checkpoint_path)
writer.close()
fig, axs = plt.subplots(2, 2, figsize=(12, 8))
epochs = list(range(len(train_loss_list)))
# Plot training loss
axs[0, 0].plot(train_loss_list)
axs[0, 0].set_title('Training Loss')
axs[0, 0].set_xlabel('Epoch')
# Plot validation loss
axs[0, 1].plot(loss_list)
axs[0, 1].set_title('Validation Loss')
axs[0, 1].set_xlabel('Epoch')
# Plot validation IoU
axs[1, 0].plot(iou_list)
axs[1, 0].set_title('Validation IoU')
axs[1, 0].set_xlabel('Epoch')
# Plot validation F1
axs[1, 1].plot(f1_list)
axs[1, 1].set_title('Validation F1')
axs[1, 1].set_xlabel('Epoch')
plt.tight_layout()
# Save the figure
plt.savefig(f'{exp_save_dir}/{args.model}_{args.train_file_dir}_{args.batch_size}_{args.max_epochs}_{args.seed}_{args.base_lr}_{train_metric_dict["best_iou"]:.4f}.png')
train_metric_dict=convert_to_numpy(train_metric_dict)
logger.info(f"Training completed. Best IoU: {train_metric_dict['best_iou']}, Best Epoch: {train_metric_dict['best_epoch']}, Best SE: {train_metric_dict['best_iou_withSE']}, Best PC: {train_metric_dict['best_iou_withPC']}, Best F1: {train_metric_dict['best_iou_withF1']}, Best ACC: {train_metric_dict['best_iou_withACC']}")
logger.info(f"Last IoU: {train_metric_dict['last_iou']}, Last SE: {train_metric_dict['last_SE']}, Last PC: {train_metric_dict['last_PC']}, Last F1: {train_metric_dict['last_F1']}, Last ACC: {train_metric_dict['last_ACC']}")
return train_metric_dict
if __name__ == "__main__":
print(f"\n=== Testing model: {args.model} ===")
exp_save_dir, writer, logger, model = init_dir(args)
row_data=vars(args)
if args.just_for_test:
if args.zero_shot_dataset_name != "":
csv_file = f"./result/result_{args.dataset_name}_2_{args.zero_shot_dataset_name}_test.csv"
else:
csv_file = f"./result/result_{args.dataset_name}_test.csv"
file_exists = os.path.isfile(csv_file)
model, model_path = load_model(args, model_best_or_final="best")
print(f"Just for test, skipping training. loading model form best checkpoint. Model loaded from {model_path}")
val_metric_dict = validate(args,logger, model)
if args.zero_shot_dataset_name !="":
zeroshot_result=zero_shot(args,logger, model)
else:
zeroshot_result=None
if val_metric_dict:
row_data.update(val_metric_dict)
if zeroshot_result:
row_data.update(zeroshot_result)
with open(csv_file, 'a', newline='') as f:
writer = csv.DictWriter(f, fieldnames=row_data.keys())
if not file_exists:
writer.writeheader()
writer.writerow(row_data)
exit()
try:
csv_file = f"./result/result_{args.dataset_name}_train.csv"
file_exists = os.path.isfile(csv_file)
train_metric_dict = train(args,exp_save_dir, writer, logger, model)
if args.zero_shot_dataset_name !="":
zeroshot_result=zero_shot(args,logger, model)
else:
zeroshot_result=None
if train_metric_dict:
row_data.update(train_metric_dict)
if zeroshot_result:
row_data.update(zeroshot_result)
with open(csv_file, 'a', newline='') as f:
writer = csv.DictWriter(f, fieldnames=row_data.keys())
if not file_exists:
writer.writeheader()
writer.writerow(row_data)
print(f"Model {args.model} tested successfully")
except Exception as e:
row_data.update({"Error": str(e)})
error_row = row_data.copy()
with open('./ERROR.log', 'a', newline='') as f:
writer = csv.DictWriter(f, fieldnames=error_row.keys())
if not file_exists:
writer.writeheader()
writer.writerow(error_row)
print(f"Model {args.model} failed: {str(e)}")
print(f"Model {args.model} tested successfully")
版权说明:
本文由 youcans@xidian 对论文 “U-Bench: 基于 100 种变体基准测试的 U-Net 全面解析”(U-Bench: A comprehensive understanding of U-Net through 100-variant benchmarking) 进行摘编和翻译。该论文版权属于原文期刊和作者,本译文只供研究学习使用。
引用格式: Tang, F., Dong, C., Ma, W., et al. (2025). U-Bench: A comprehensive understanding of U-Net through 100-variant benchmarking. arXiv preprint arXiv:2510.07041.
youcans@xidian 作品,转载必须标注原文链接:
U-Bench: 基于 100 种变体基准测试的 U-Net 全面解析
(https://youcans.blog.csdn.net/article/details/154533776)
Crated:2025-11

火山引擎开发者社区是火山引擎打造的AI技术生态平台,聚焦Agent与大模型开发,提供豆包系列模型(图像/视频/视觉)、智能分析与会话工具,并配套评测集、动手实验室及行业案例库。社区通过技术沙龙、挑战赛等活动促进开发者成长,新用户可领50万Tokens权益,助力构建智能应用。
更多推荐
 已为社区贡献45条内容
已为社区贡献45条内容






所有评论(0)